清华新闻网6月27日电 目前绝大多数的临床药物以蛋白质作为靶标,然而,许多蛋白质由于缺乏合适的结构口袋,常被认为是“难成药”或“不可成药”的。在人类的2万个左右的蛋白编码基因(占人类基因组总长度的1.5%左右)中,大约有10%至15%与疾病直接相关;而在这些基因中,据估计仅有700至900个的蛋白产物是可以成药的(仅占人类基因组总长度的0.05%左右)。另一方面,人类基因组的约70%甚至更多都会被转录成RNA,其中大多是非编码RNA(ncRNA)。因此,近年来开始有越来越多的研究者试图将RNA作为药物靶标,并初步证明了这一策略的可行性。新药研发成本昂贵且周期漫长,使用计算机辅助药物设计能够极大地降低研发成本,助力并加速靶向RNA的小分子药物的研发进程。然而,由于公开的RNA-小分子互作及已知的高分辨率RNA结构数据匮乏,开发数据驱动的深度学习模型仍面临诸多挑战。
6月24日,清华大学生命学院鲁志课题组和合作者在《自然·计算科学》(Nature Computational Science)发表了题为“通过数据扰动与增强模型进行RNA-配体相互作用预测”(RNA-ligand interaction scoring via data perturbation and augmentation modeling)的研究论文。该研究突破了传统药物设计方法依赖三维结构的局限,针对RNA结构匮乏的现实,提出了一种基于序列输入的RNA-小分子相互作用AI预测模型RNAsmol。该模型为靶向RNA的小分子药物研发提供了高效计算工具,为不依赖三维结构的人工智能辅助药物设计提供了新的方案和思路。
该工作提出的RNAsmol框架,是一种结合数据扰动和数据增广策略的深度学习方法。在该框架中,数据扰动通过对训练数据进行随机扰动模拟真实环境中的数据多样性,从而帮助模型更好地学习到RNA-小分子结合的规律。而数据增广则通过在已知互作的基础上生成虚拟的负样本和潜在的未标记样本,增加模型对未知空间的识别能力。这种策略不仅提高了模型的鲁棒性,还帮助其更好地捕捉到不同类型的相互作用模式。此外,该模型结合基于图的分子特征表示方法和图扩散卷积模块对药物小分子进行结构建模,通过基于注意力机制的特征融合模块在多个模态下对靶标和药物分子特征进行加权整合,最终实现RNA靶标与小分子之间的打分预测。
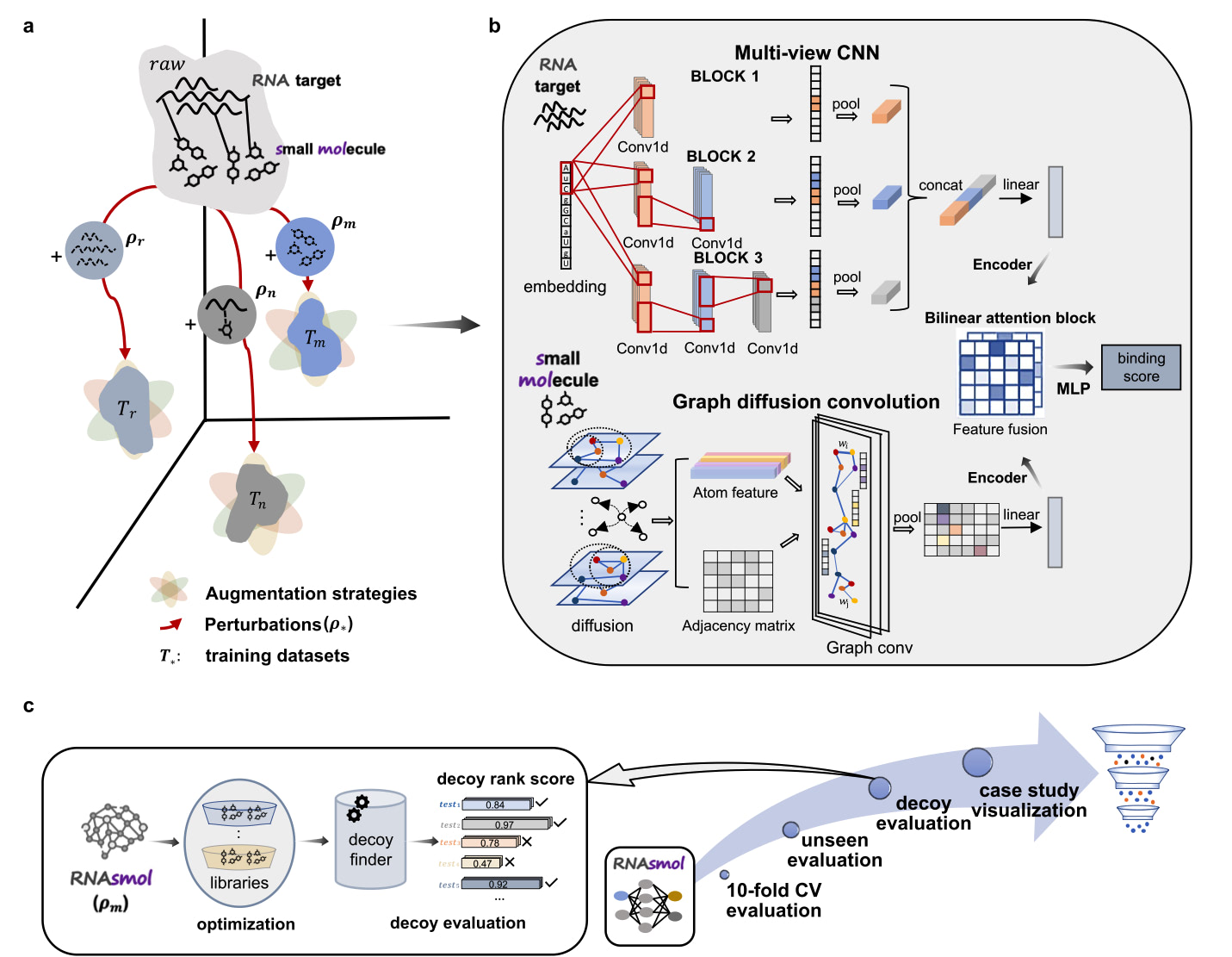
图1.RNAsmol模型和整体计算框架
在数据扰动空间中,RNAsmol通过扰动策略有效地减少了真实负样本与未知互作空间之间的偏差。该策略通过对已知的负样本进行扰动,生成潜在的“负例”样本,同时通过数据增广技术扩展已知的正例和负例样本的边界。这使得模型能够更好地理解RNA与小分子之间的结合规律,尤其是在数据不均衡的情况下,避免了模型偏向已知的正负样本。实验结果表明,RNAsmol在10折交叉验证中的表现超过了传统方法,平均AUROC(曲线下面积)指标提升约8%,同时在未见样本的评估中,性能提升了约16%。这一优势证明了该方法在稀疏数据场景中的有效性,进一步推动了RNA-小分子结合预测的计算研究。
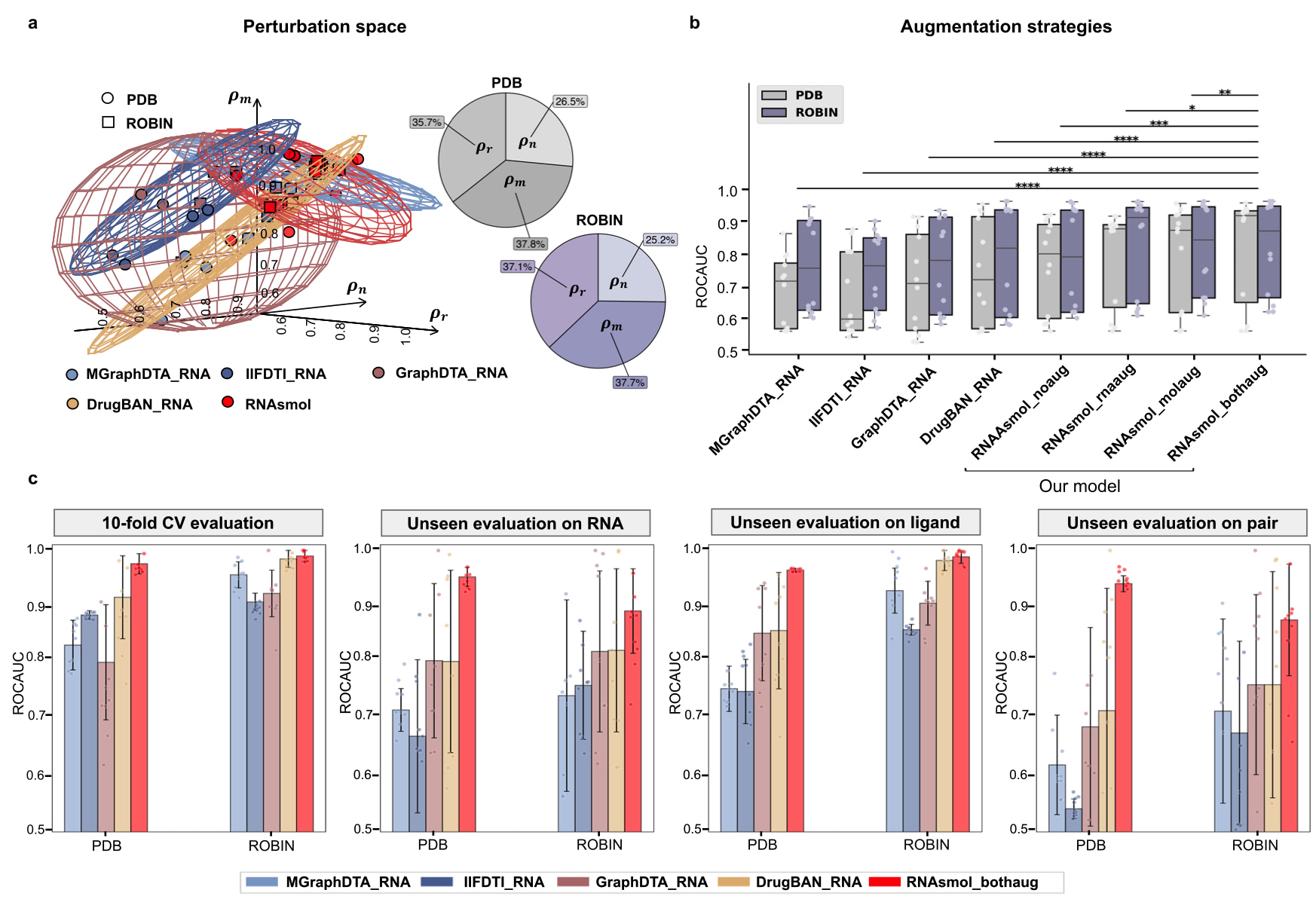
图2.RNAsmol在不同验证集上的预测效果评估
在虚拟筛选的应用中,RNAsmol展现出独特的优势。与传统依赖于结构信息的筛选方法不同,RNAsmol完全基于RNA的序列信息进行预测。因为许多与疾病相关的RNA靶点(如lncRNA)的三维结构数据常常难以获得,RNAsmol能够弥补这一数据缺口实现对这些靶点的预测筛选。实验结果表明,RNAsmol在区分诱饵分子与真实配体时,成功将排序得分提高了约30%。因此,RNAsmol在各类RNA靶向药物筛选中具有广泛的适用性,通过该方法可以更加高效地筛选潜在的药物分子。
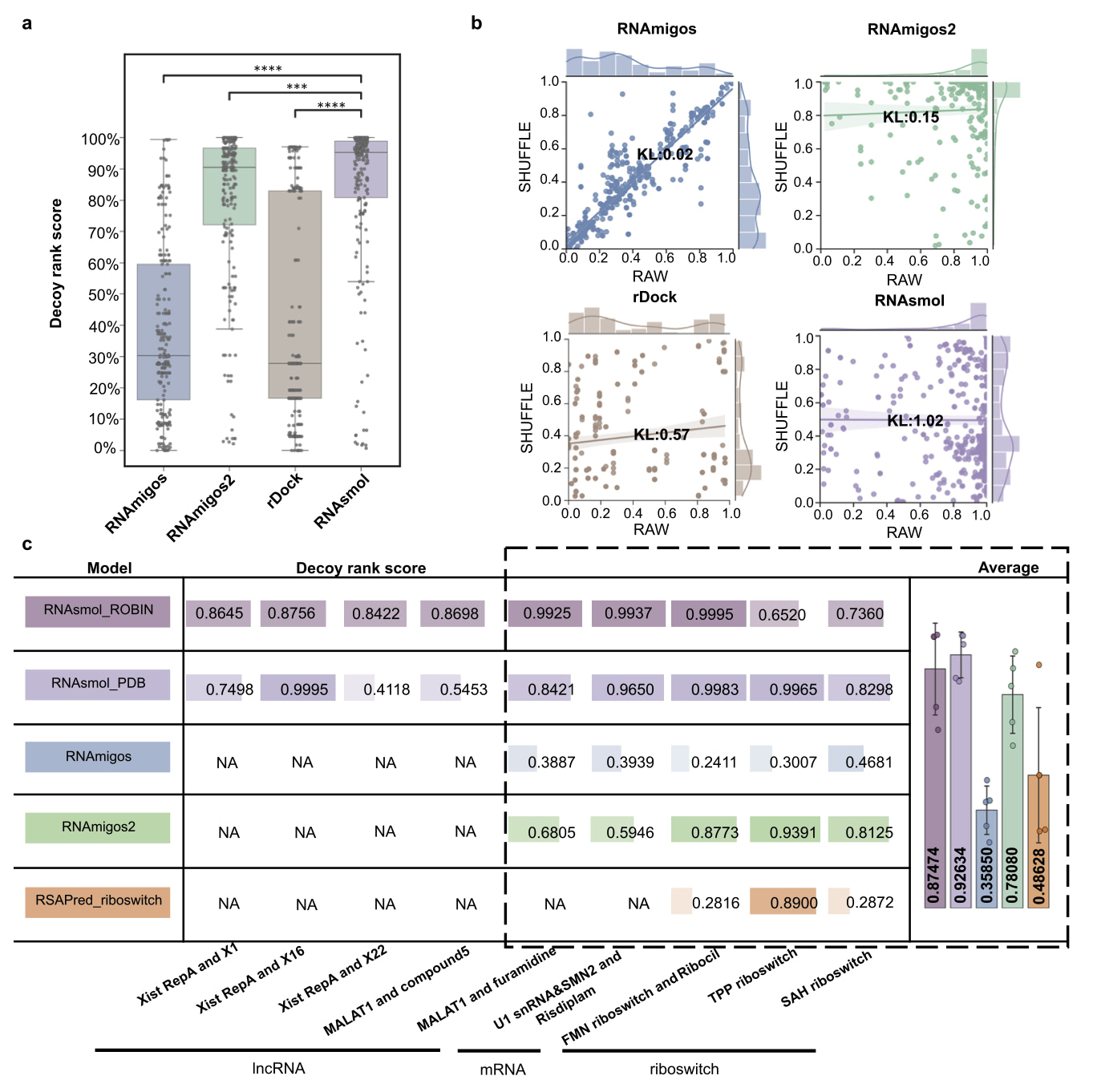
图3.RNAsmol和其他计算方法的比较
综上所述,研究通过探究基于数据扰动和增广的深度学习训练策略在数据匮乏场景的应用,为靶向RNA药物研发的计算建模提供了新思路。
清华大学生命学院副教授鲁志与南昌大学教授徐振江为论文通讯作者。清华大学已出站博士后(现为哈尔滨工业大学副研究员)马洪丽为论文第一作者。研究得到国家重点研发计划、国家自然科学基金、“生物信息学”教育部重点实验室、“绿色生物制造”全国重点实验室、清华大学精准医学研究院、拜耳公司等的资助支持。
论文链接:
https://www.nature.com/articles/s43588-025-00820-x
供稿:生命学院
编辑:李华山
审核:郭玲